期刊:Nature Communation
影响因子:12.124
主要技术:CRISPR-Cas9
研究背景
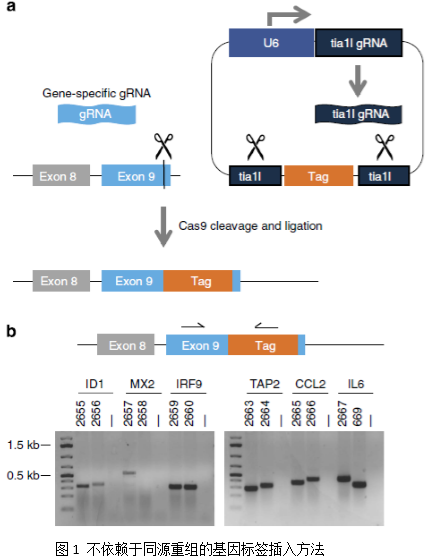
CRISPR-Cas9技术虽然使基因敲除变得简便,但标签插入依然由于每插入一个位点都需要针对该位点构建donor质粒且依赖自发的同源重组而显得昂贵且存在一定难度。本研究发现了一种能解决上述问题的基因标签插入方法,该方法只需构建一个donor质粒即可用于所有位点的精准的标签插入且不依赖于同源重组。
研究内容及结果
1. 本研究首先构建了能表达斑马鱼tia1l基因sgRNA的donor质粒,该质粒在Cas9蛋白的存在下能释放杀稻瘟菌素CDS,与携带Cas9蛋白和特定位点sgRNA的质粒共转入大多数基因都只有1个拷贝的HAP1细胞中,初步评估了杀稻瘟菌素CDS插入的效率,6个基因12个位点中有11个成功插入了杀稻瘟菌素CDS。

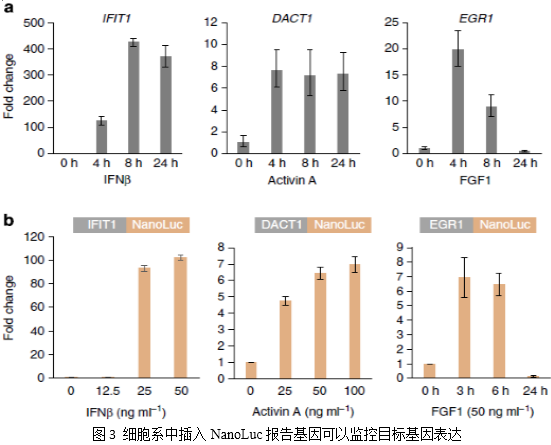
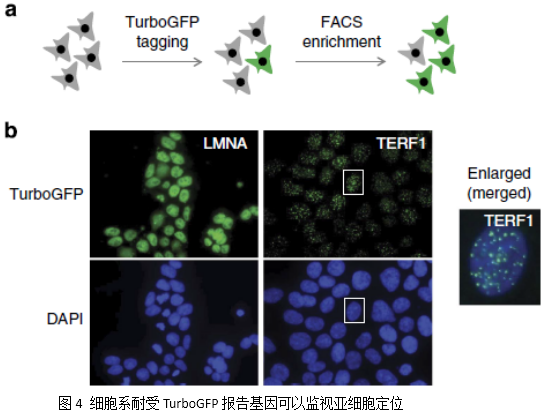
运用本研究中的标签插入方法,可以对目标细胞系靶基因定点插入NanoLuc、TurboGFP或其他标签,实现对靶基因表达量的监控或进行亚细胞定位。该方法比传统的依赖于同源重组双交换的标签插入方法更加省时,操作上更为便捷,且一个donor载体可以用于不同基因不同细胞的同一种标签插入,节约成本。
解析文献
Daniel H. Lackner, Alexia Carre´, et al. A generic strategy for CRISPR-Cas9-mediated gene tagging [J]. Nature Communications,2015,6:10237.
参考文献
1.Auer, T. O., Duroure, K., De Cian, A., Concordet, J. P. & Del Bene, F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homologyindependent DNA repair. Genome Res. 24, 142–153 (2014).
2. Maresca, M., Lin, V. G., Guo, N. & Yang, Y. Obligate ligation-gated recombination (ObLiGaRe): custom-designed nuclease-mediated targeted integration through nonhomologous end joining. Genome Res. 23, 539–546(2013).
3. Cristea, S. et al. In vivo cleavage of transgene donors promotes nucleasemediated targeted integration. Biotechnol. Bioeng. 110, 871–880 (2013).
4. Ran, F. A. et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature 520, 186–191 (2015).
5. Cong, L. et al. Multiplex genome engineering using CRISPR/Cas systems. Science 339, 819–823 (2013).
